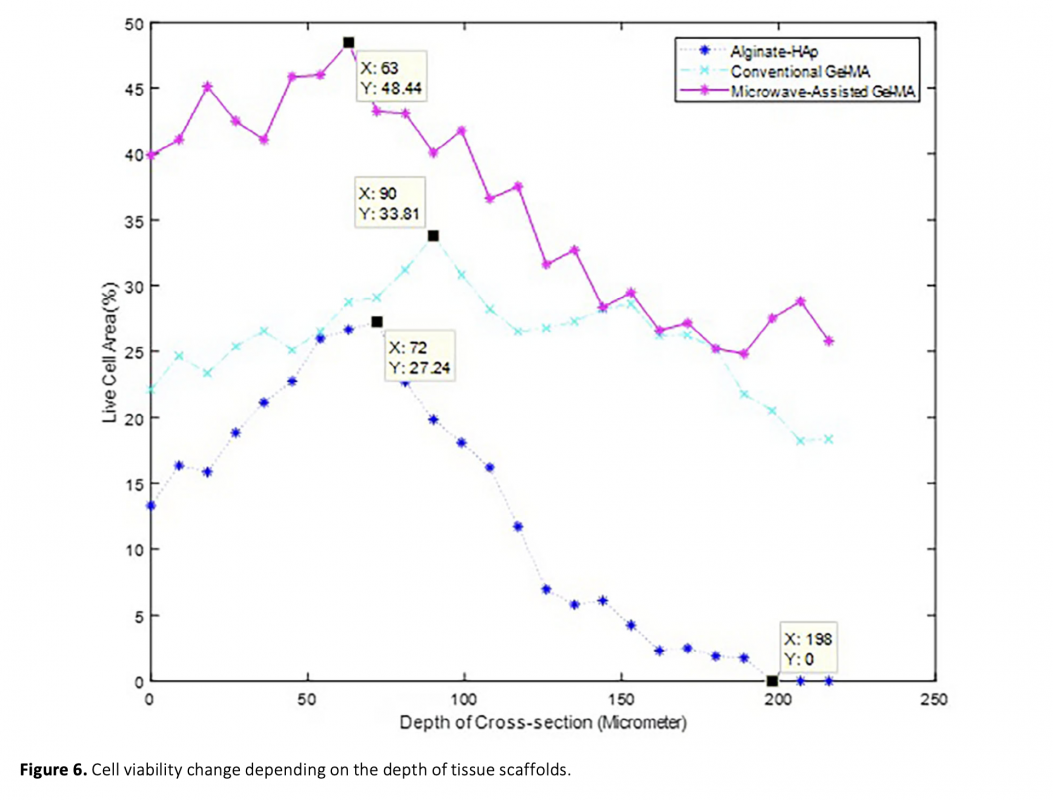
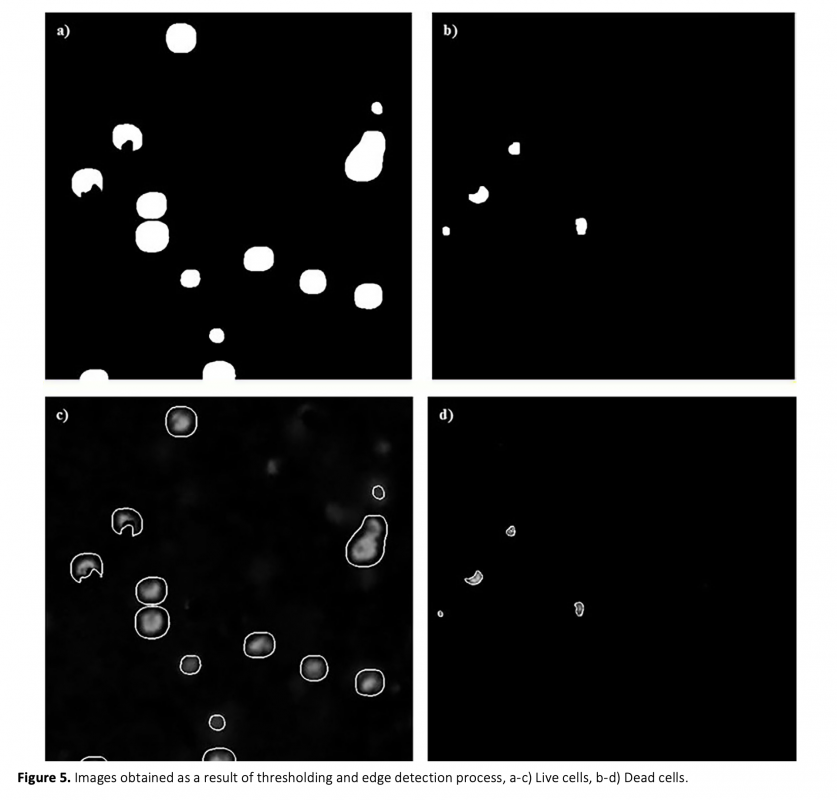
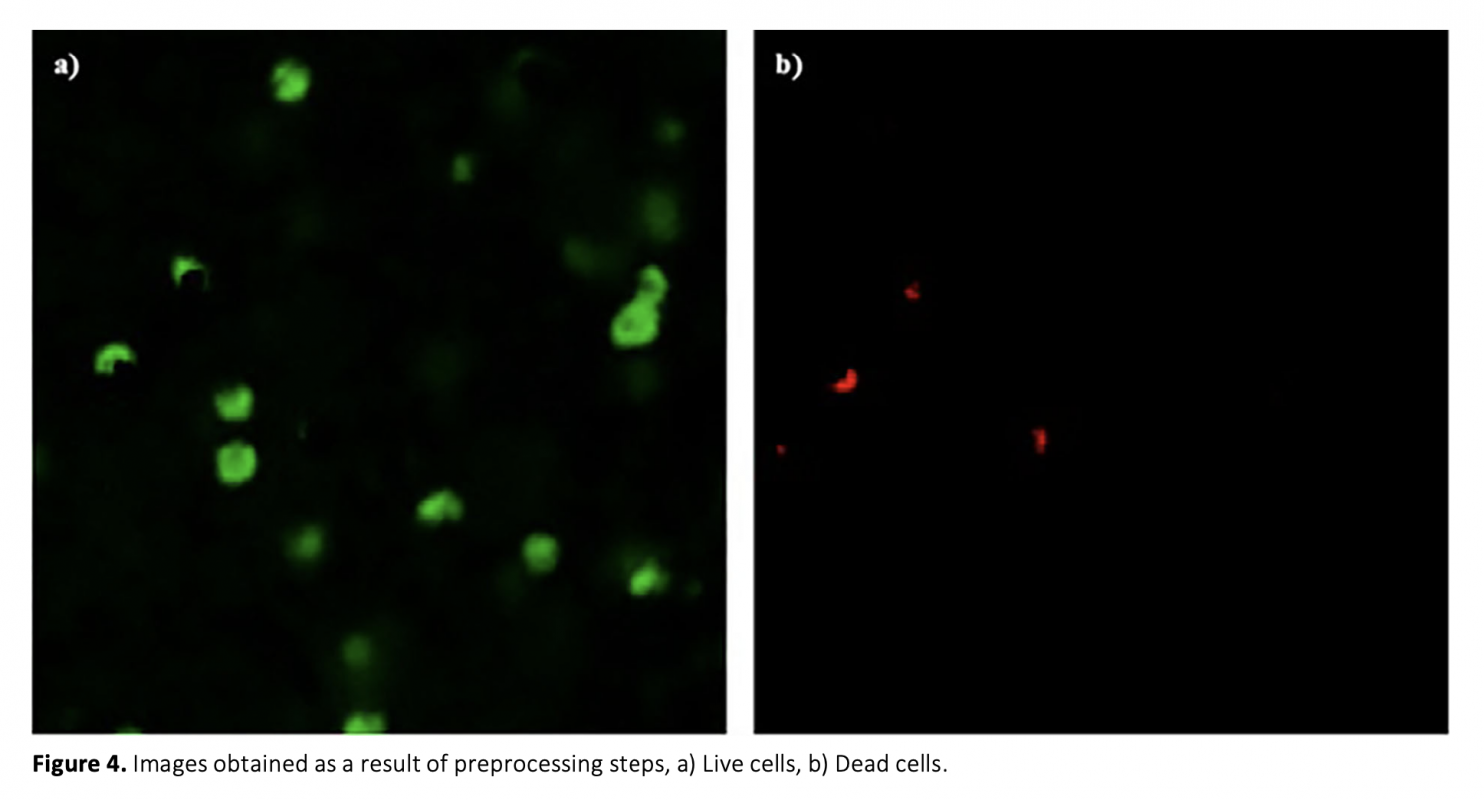
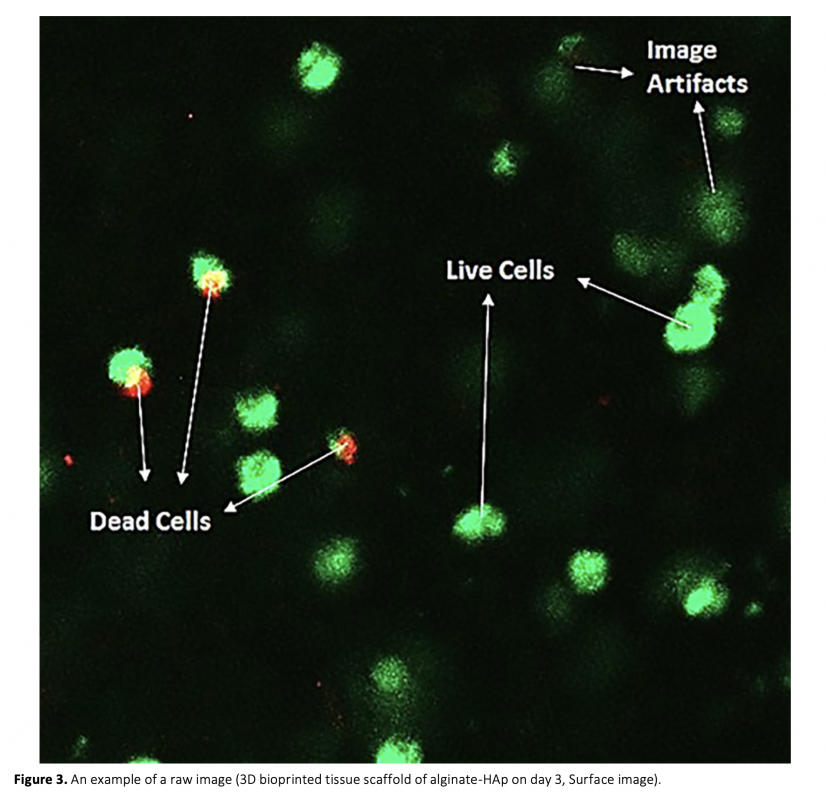
Image analysis of cell biology and tissue engineering is time-consuming and requires personal expertise. However, evaluation of the results may be subjective. Therefore, computer-based learning and detection applications have been rapidly developed in recent years. In this study, Confocal Laser Scanning Microscope (CLSM) images of the viable pre-osteoblastic mouse MC3T3-E1 cells in 3D bioprinted tissue scaffolds, captured from a bone tissue regeneration study, were analyzed by using image processing techniques. The aim of this study is to develop a reliable and fast algorithm for the automated analysis of live/dead assay CLSM images. Percentages of live and dead cell areas in the scaffolds were determined, and then, total cell viabilities were calculated. Furthermore, manual measurements of four different analysts were obtained to evaluate subjectivity in the analysis. The measurement variations of analysts, also known as the coefficient of variation, were determined from 13.18% to 98.34% for live cell images and from 9.75% to 126.02% for dead cell images. Therefore, an automated algorithm was developed to overcome this subjectivity. The other aim of this study is to determine the depth profile of viable cells in 3D tissue scaffolds. Consequently, cross-sectional image sets of three different types of tissue scaf- folds were analyzed.
Hücre biyolojisi ve doku mühendisliğinde görüntü analizi zaman alan ve kişisel uzmanlık gerektiren bir işlemdir. Ancak sonuçların değerlendirilmesi öznel olabilir. Bu nedenle bilgisayar destekli öğrenme ve tespit uygulamaları son yıllarda hızla gelişmiştir. Bu çalışmada, bir kemik dokusu rejenerasyon çalışmasından yakalanan, 3D biyo-baskılı doku iskelelerinde canlı pre-osteoblastik fare MC3T3-E1 hücrelerinin Konfokal Lazer Taramalı Mikroskop (CLSM) görüntüleri, görüntü işleme teknikleri kullanılarak analiz edilmiştir. Bu çalışmanın amacı, canlı/ölü analizi CLSM görüntülerinin otomatik analizi için güvenilir ve hızlı bir algoritma geliştirmektir. İskelelerdeki canlı ve ölü hücre alanlarının yüzdeleri belirlenmiş ve ardından toplam hücre canlılıkları hesaplanmıştır. Ayrıca, analizde öznelliği değerlendirmek için dört farklı analistin manuel ölçümleri yapılmıştır. Varyasyon katsayısı olarak da bilinen analistlerin ölçüm varyasyonları, canlı hücre görüntüleri için % 13.18 ile % 98.34 ve ölü hücre görüntüleri için % 9.75 ile % 126.02 arasında belirlenmiştir. Bu nedenle, bu öznelliği aşmak için otomatik bir algoritma geliştirilmiştir. Bu çalışmanın diğer amacı, 3 boyutlu doku iskelelerindeki canlı hücrelerin derinlik profilini belirlemektir. Sonuç olarak, üç farklı doku iskelesinin kesitsel görüntü setleri analiz edilmiştir.
Download Article in PDF (3.2 MB)